AlphaFold 2 di Google risolve uno dei più grandi problemi della biologia, il ripiegamento delle proteine
di Riccardo Robecchi pubblicata il 07 Dicembre 2020, alle 09:31 nel canale Innovazione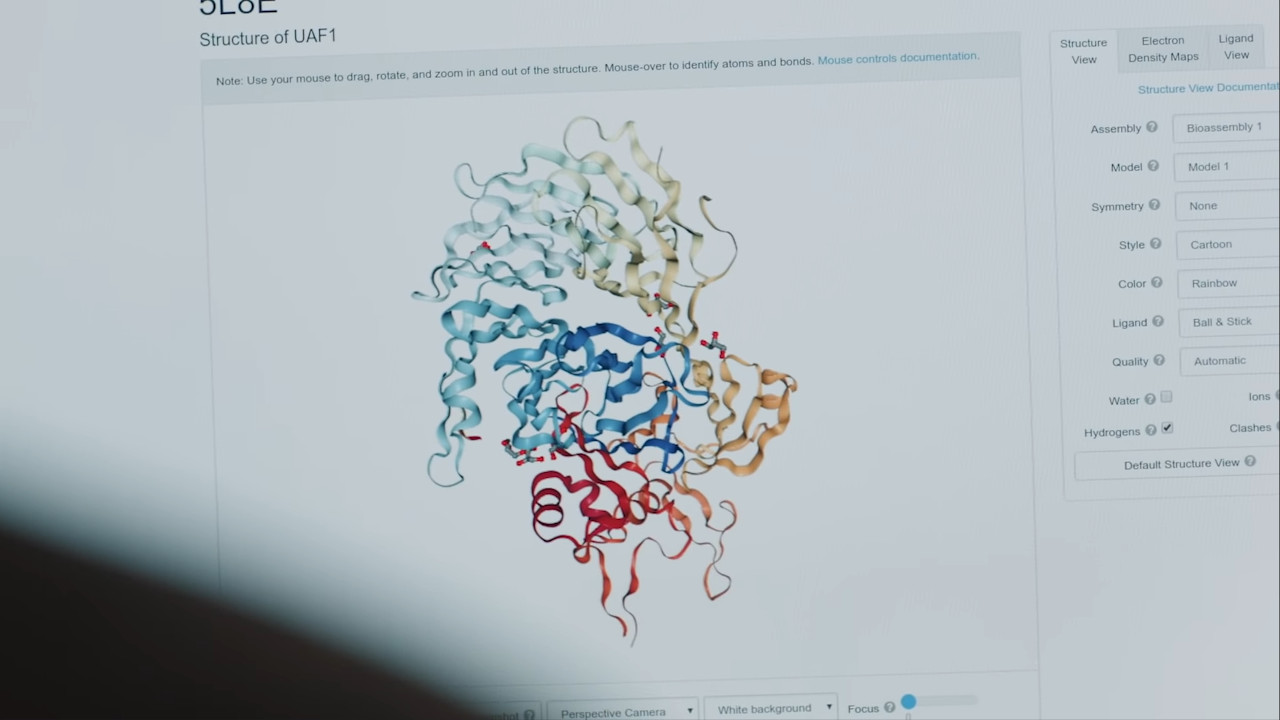
AlphaFold 2 è l'intelligenza artificiale sviluppata da DeepMind, parte della famiglia di Google, che ha risolto per la prima volta il problema del ripiegamento delle proteine in maniera convincente. Un passo in avanti notevole per la biologia
Si chiama AlphaFold 2 ed è potenzialmente uno dei più grandi passi avanti nella biologia degli ultimi 50 anni. L'intelligenza artificiale di DeepMind, nata all'interno di Google e ora sotto il cappello di Alphabet, ha infatti risolto uno dei più grandi e vecchi problemi della biologia: prevedere la forma delle proteine una volta ripiegate. Il problema del ripiegamento delle proteine ha più di cinquant'anni e vede ora una possibile soluzione che potrà portare, ad esempio, a sviluppare nuovi farmaci e a capire meglio come evolvano le cellule tumorali.
Il ripiegamento delle proteine: un modello estremamente complesso
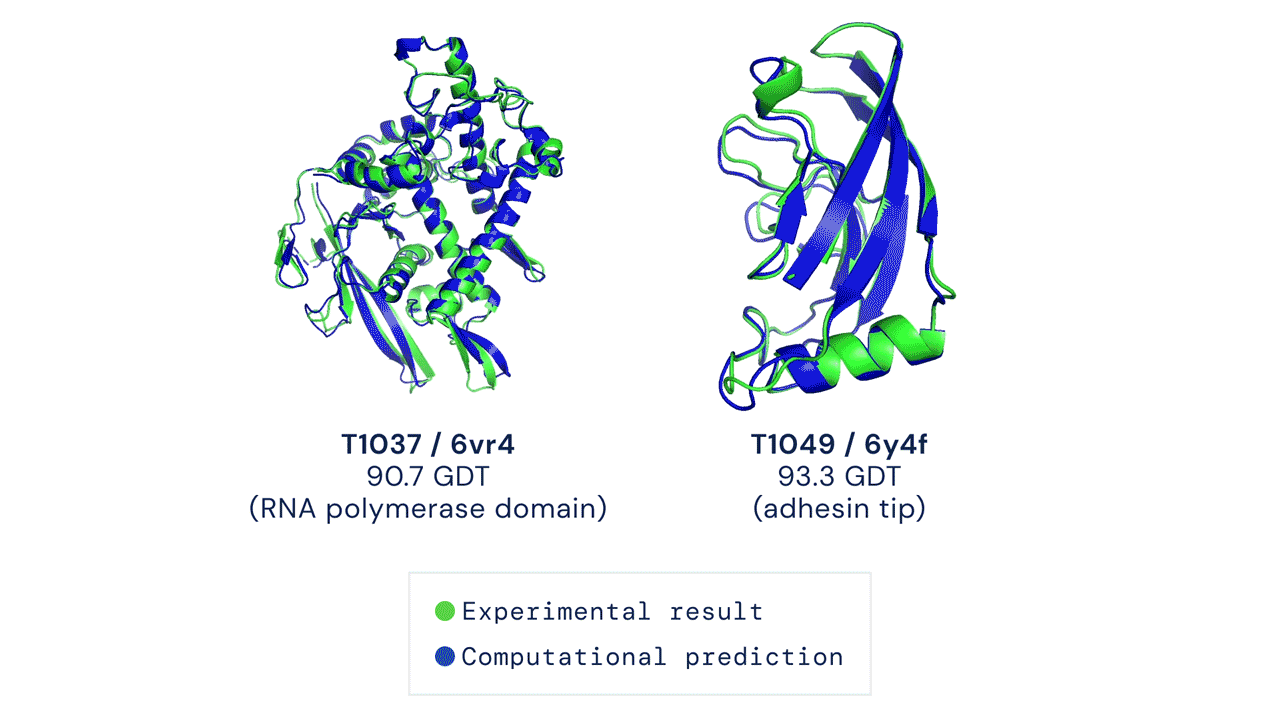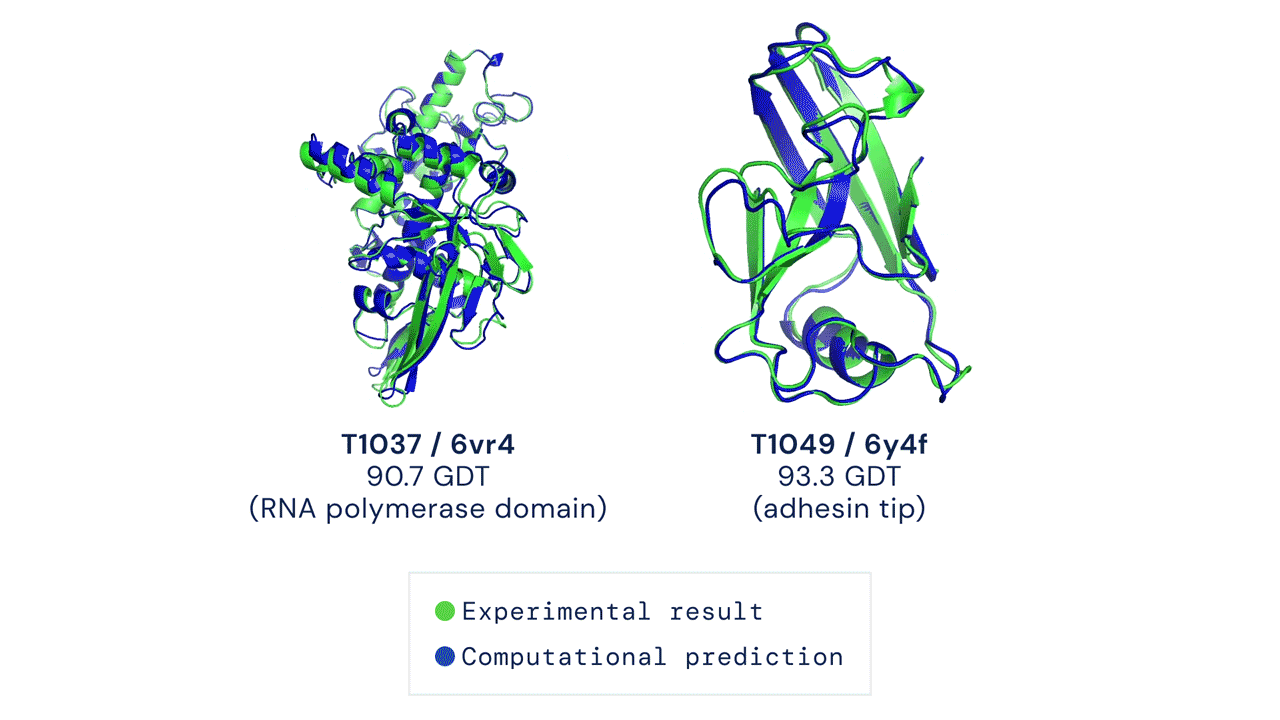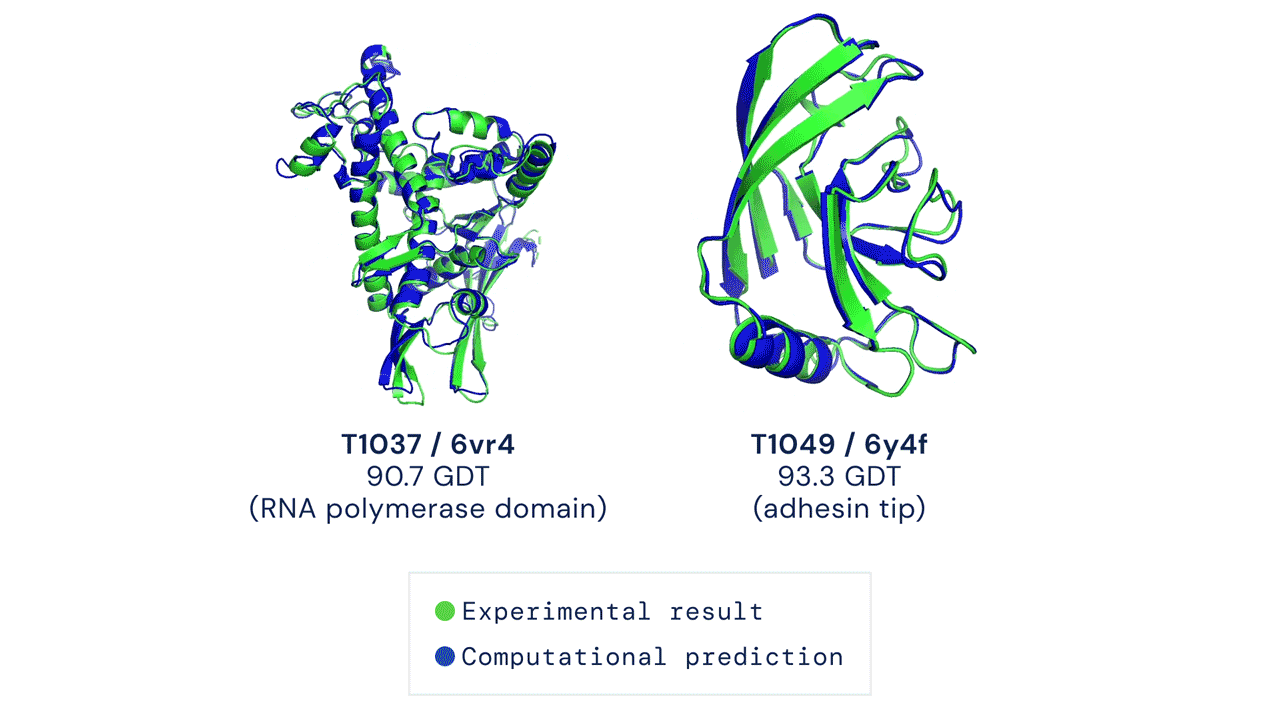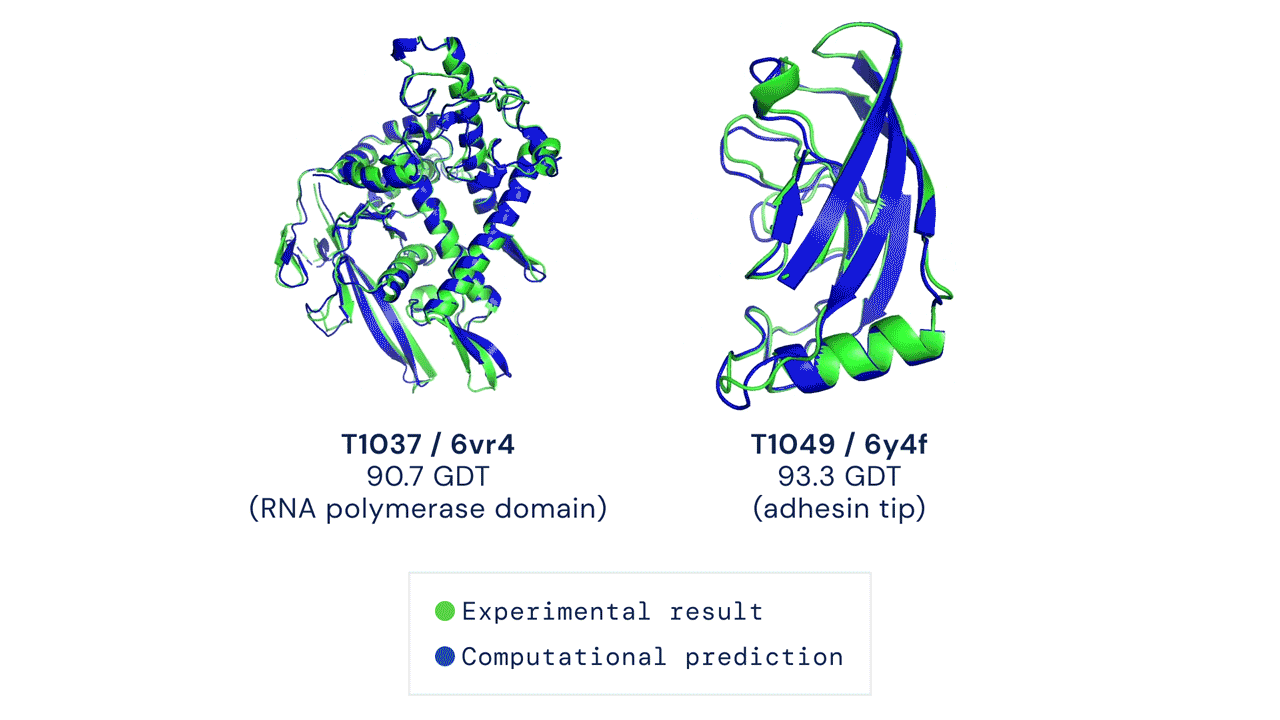
Sebbene vengano rappresentate come catene di atomi bidimensionali, le proteine assumono forme tridimensionali quando vengono sintetizzate dalle cellule. Le forme assunte dalle proteine ne determinano le caratteristiche e, quindi, conoscerle in anticipo significa poter predire anche le proprietà di una certa proteina e come interagirà con altri composti e con le cellule. Le applicazioni in ambito medico sono quindi molteplici.
All'epoca della formulazione del problema, nel 1969, Cyrus Levinthal stimò che i possibili ripiegamenti per una proteina media fossero nell'ordine di 10³⁰⁰ (1 seguito da 300 zeri), una quantità tale che si stimava potesse essere calcolata solo in un tempo superiore all'età dell'Universo conosciuto. Eppure le proteine si ripiegano spontaneamente e talora nell'arco di pochi millisecondi: una apparente incongruenza nota come paradosso di Levinthal.
Il premio Nobel per la chimica nel 1972, Christian Anfinsen, aveva tenuto il suo discorso di accettazione del premio postulando che la sequenza degli amminoacidi che compongono una proteina dovrebbe, in teoria, permettere di determinarne interamente la struttura. Proprio tale concetto ha portato alla sfida di calcolare la forma delle proteine una volta ripiegate: una sfida che ha visto anche l'istituzione di una sfida formale, la CASP (Critical Assessment of protein Structure Prediction, ovvero valutazione critica della predizione della struttura delle proteine).
La CASP è stata fondata nel 1994 e assegna ogni due anni un punteggio da 0 a 100 ai metodi di predizione. Il punteggio rappresenta la percentuale di amminoacidi entro la distanza di tolleranza dalla posizione corretta (GDT, Global Distance Test) ed è dunque una misura reale dell'accuratezza del metodo di previsione. Per assicurarsi che i metodi siano testati in maniera veritiera, viene chiesto dagli organizzatori di prevedere la struttura tridimensionale di proteine di cui non sia ancora stata pubblicata la misura sperimentale - in altri termini, non sono state pubblicate informazioni su come la proteina sia fatta nella realtà. Perché un modello di predizione sia considerato efficace deve superare il punteggio di 90, considerato competitivo con le misure sperimentali.
L'intelligenza artificiale AlphaFold 2 risolve il problema del ripiegamento delle proteine
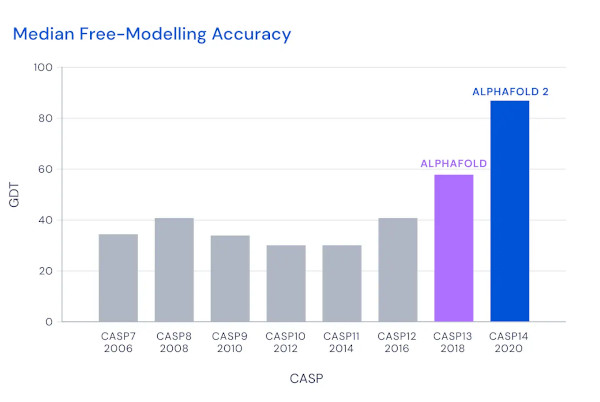
DeepMind afferma di aver risolto il problema durante CASP14, la quattordicesima edizione che si tiene quest'anno, arrivando a un punteggio di 92,4 GDT. Lo scorso anno, la prima generazione dell'intelligenza artificiale AlphaFold aveva raggiunto un punteggio di 60.
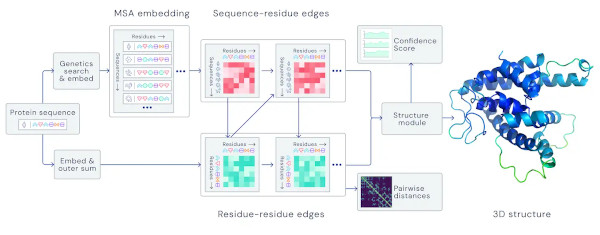
"Per l'ultima versione di AlphaFold, utilizzata per CASP14, abbiamo creato un sistema di rete neurale basato sull'attenzione, addestrato end-to-end, che tenta di reinterpretare la struttura del grafo [che rappresenta la struttura della proteina] mentre ragiona sul grafo implicito che sta costruendo. Usa sequenze correlate evolutivamente, allineamento di sequenze multiple e una rappresentazione delle coppie di residui di amminoacidi per raffinare questo grafo", scrivono in un articolo sul blog di DeepMind i ricercatori autori di AlphaFold 2. "Iterando questo processo, il sistema sviluppa predizioni forti della struttura proteica sottostante ed è quindi in grado di determinare strutture molto accurate nel giro di giorni. In più, AlphaFold può predire quali parti di ciascuna struttura proteica predetta siano affidabili con una misura interna della confidenza."
DeepMind ha raggiunto questo risultato addestrando la propria intelligenza artificiale con metodi di machine learning usando circa 170.000 strutture proteiche ottenute da database pubblici assieme a sequenze di proteine la cui struttura non è ancora nota. Il sistema utilizza 16 TPUv3 (Tensor Processing Unit, un ASIC specifico per l'IA sviluppato da Google), equivalenti a circa 100 o 200 GPU.
Quale impatto avrà AlphaFold 2 sulla biologia e la medicina?
Poter predire con certezza la struttura proteica potrà portare a una migliore comprensione di come funzionino le nostre cellule e, di conseguenza, a poter sviluppare farmaci più efficaci o che siano in grado di curare malattie per cui ora non esistono cure. È difficile fare predizioni accurate di come l'intelligenza artificiale AlphaFold 2 potrà aiutare i ricercatori, ma uno degli ambiti di maggiore interesse è quello della ricerca sul cancro: molte sue forme infatti hanno a che fare con proteine malfunzionanti.
I ricercatori mettono le mani avanti: "AlphaFold è uno dei nostri risultati più grandi finora ma, come con tutta la ricerca scientifica, ci sono ancora molte domande a cui rispondere. Non tutte le strutture che prediremo saranno perfette. [...] Ma come 50 anni fa, quando Anfinsen ha lanciato una sfida ben al di là delle capacità della scienza al tempo, ci sono molti aspetti del nostro universo che rimangono sconosciuti. Il progresso che abbiamo annunciato oggi ci dà maggiore sicurezza che l'intelligenza artificiale diventerà uno degli strumenti più utili all'umanità per espandere le frontiere della conoscenza scientifica, e non vediamo l'ora di vedere i molti anni di duro lavoro e di scoperte che abbiamo davanti!"
Rimane comunque da capire quando Google renderà disponibile il sistema alla comunità scientifica così che possa metterlo in opera per ragiungere nuove scoperte rilevanti.











 Ryzen Threadripper 9980X e 9970X alla prova: AMD Zen 5 al massimo livello
Ryzen Threadripper 9980X e 9970X alla prova: AMD Zen 5 al massimo livello Acer TravelMate P4 14: tanta sostanza per l'utente aziendale
Acer TravelMate P4 14: tanta sostanza per l'utente aziendale Hisense M2 Pro: dove lo metti, sta. Mini proiettore laser 4K per il cinema ovunque
Hisense M2 Pro: dove lo metti, sta. Mini proiettore laser 4K per il cinema ovunque Xbox: dal prossimo mese verifica dell'identità obbligatoria in UK, in futuro anche in altre regioni
Xbox: dal prossimo mese verifica dell'identità obbligatoria in UK, in futuro anche in altre regioni Corsair AI Workstation 300: poco più grande di un Mini PC, ma con la potenza di un desktop
Corsair AI Workstation 300: poco più grande di un Mini PC, ma con la potenza di un desktop Addio a Elio Marioli, un pioniere della mobilità elettrica quando nessuno ne parlava
Addio a Elio Marioli, un pioniere della mobilità elettrica quando nessuno ne parlava








21 Commenti
Gli autori dei commenti, e non la redazione, sono responsabili dei contenuti da loro inseriti - infoCknsknskjnbslmkns
Quindi in buona sostanza adesso non serve più?
E' sempre il progetto ROSETTA.
E' stata fatta molta ricerca ed enormi quantità di simulazioni.. che non implica arrivare necessariamente ad un traguardo purtroppo.
Comunque molti dei risultati ottenuti da queste simulazioni probabilmente sono stati usati anche per lo sviluppo dei vaccini che stanno arrivnado ora.
Assolutamente no.
Folding si occupa non solo di previsione di proteine, ma anche della loro interazione con altre proteine (tecnicamente si chiama "docking"
Quanto ottenuto da AlphaFold è incredibile, ma è solo l'inizio!!
Quindi in buona sostanza adesso non serve più?
Come sopra, no.
Rosetta (come Folding) è una piattaforma di simulazioni eterogenee (ab_initio, folding, docking, ecc). Quello di AlphaFold è solo l'inizio (incredibile) di una fantastica avventura.
P.S. Il team di Rosetta e quello di AlphaFold, in passato, hanno già collaborato e collaboreranno ancora, introducendo nel codice di Rosetta parti di AlphaFold e viceversa.
AI, reti neurali profonde e, in un futuro abbastanza lontano (ma non troppo) server quantistici, rivoluzioneranno letteralmente l'approccio alla biologia (e alla medicina).
Togli il "probabilmente".
Affermazioni analoghe a quelle di qualche decina di anni fa con l'avvento dei personal computer, e siamo finiti a pubblicare video di gattini e a dare voce a gente che crede che la terra sia piatta e che da fama mondiale a ragazzine che fanno smorfie in video di dieci secondi...
Devi effettuare il login per poter commentare
Se non sei ancora registrato, puoi farlo attraverso questo form.
Se sei già registrato e loggato nel sito, puoi inserire il tuo commento.
Si tenga presente quanto letto nel regolamento, nel rispetto del "quieto vivere".